QS Edizioni - giovedì 21 novembre 2024
Scienza e Farmaci
Genoma umano. Su Science lo studio con la mappa completa che svela l’8% ancora mancante
di Will Dunham- allegati(1)
 1 aprile - Pubblicato ieri sulla rivista americana il primo genoma umano completo, colmando così le lacune rimanenti e offrendo nuove promesse nella ricerca di indizi sulle mutazioni che causano malattie e sulla variazione genetica tra i 7,9 miliardi di persone nel mondo. LO STUDIO.
1 aprile - Pubblicato ieri sulla rivista americana il primo genoma umano completo, colmando così le lacune rimanenti e offrendo nuove promesse nella ricerca di indizi sulle mutazioni che causano malattie e sulla variazione genetica tra i 7,9 miliardi di persone nel mondo. LO STUDIO.
(Reuters) - Nel 2003 la ricerca svelava quella che fu all’epoca annunciata come la sequenza completa del genoma umano. Ma circa l'8% di esso non era stato in realtà completamente decifrato, principalmente perché consisteva in frammenti di DNA altamente ripetitivi e difficili da combinare.
Ieri un consorzio di scienziati ha colmato la lacuna e in una ricerca pubblicata sulla rivista Science ha svelato anche quell’8% mancante. Il lavoro era già stato reso pubblico lo scorso anno ma mancava la sua revisione tra pari e la pubblicazione ufficiale.
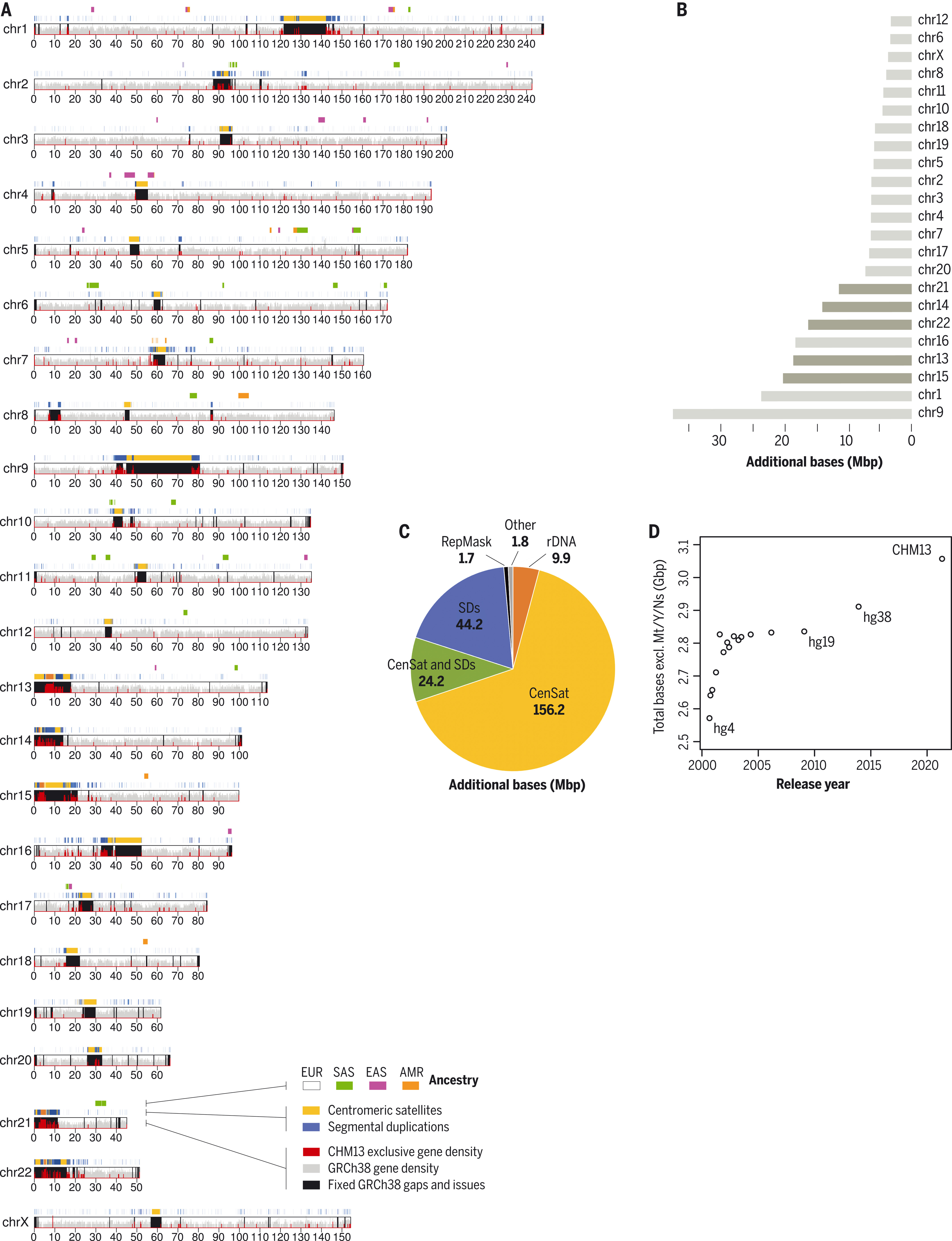
Riepilogo dell'assemblaggio completo del genoma umano T2T-CHM13.
( A ) Ideogramma delle caratteristiche dell'assieme T2T-CHM13v1.1. Per ogni cromosoma (chr), vengono fornite le seguenti informazioni dal basso verso l'alto: lacune e problemi in GRCh38 risolti da CHM13 sovrapposti con la densità dei geni esclusivi di CHM13 in rosso; duplicazioni segmentali (SD) ( 42 ) e satelliti centromerici (CenSat) ( 30 ); e previsioni sull'ascendenza CHM13 (EUR, europea; SAS, dell'Asia meridionale; EAS, dell'Asia orientale; AMR, americana mista). La scala inferiore è misurata in Mbp. ( B e C ) Basi aggiuntive (non sinteniche) nell'assieme CHM13 relative a GRCh38 per cromosoma, con gli acrocentrici evidenziati in nero (B) e per tipo di sequenza (C). (Si noti che le annotazioni CenSat e SD si sovrappongono.) RepMask, RepeatMasker. ( D) Basi non gap totali nei rilasci del genoma di riferimento UCSC risalenti al settembre 2000 (hg4) e terminano con T2T-CHM13 nel 2021. Mt/Y/N, mitocondri, chrY e lacune. (Fonte: Science)
"La generazione di una sequenza del genoma umano veramente completa rappresenta un incredibile risultato scientifico, fornendo la prima visione completa del nostro progetto di DNA", ha affermato Eric Green, direttore del National Human Genome Research Institute (NHGRI), parte del National Institutes of Health degli Stati Uniti in una dichiarazione.
"Queste informazioni fondamentali rafforzeranno i numerosi sforzi in corso per comprendere tutte le sfumature funzionali del genoma umano, che a loro volta rafforzeranno gli studi genetici sulle malattie umane", ha aggiunto Green.
La versione completa è composta da 3.055 miliardi di coppie di basi, le unità da cui sono costruiti i cromosomi e i nostri geni, e 19.969 geni che codificano per le proteine.
Di questi geni, i ricercatori ne hanno identificati circa 2.000 nuovi. La maggior parte di questi è disabilitata, ma 115 potrebbero essere ancora attivi. Gli scienziati hanno anche individuato circa 2 milioni di varianti genetiche aggiuntive, 622 delle quali erano presenti in geni clinicamente rilevanti.
Il consorzio di scienziati che ha compiuto la ricerca è stato soprannominato Telomere-to-Telomere (T2T), dal nome delle strutture che si trovano alle estremità di tutti i cromosomi, la struttura filiforme nel nucleo della maggior parte delle cellule viventi che trasporta informazioni genetiche sotto forma di geni.
"In futuro, quando qualcuno avrà il proprio genoma sequenziato, saremo in grado di identificare tutte le varianti nel suo DNA e utilizzare tali informazioni per guidare meglio la propria assistenza sanitaria", ha detto Adam Phillippy, uno dei leader di T2T e ricercatore senior presso NHGRI.
"Completare davvero la sequenza del genoma umano è stato come indossare un nuovo paio di occhiali. Ora che possiamo vedere tutto chiaramente, siamo un passo più vicini alla comprensione di cosa significa", ha aggiunto Phillippy.
Tra le altre cose, le nuove sequenze di DNA hanno fornito nuovi dettagli sulla regione intorno a quello che viene chiamato centromero, dove i cromosomi vengono afferrati e separati quando le cellule si dividono per garantire che ogni cellula "figlia" erediti il numero corretto di cromosomi.
"Scoprire la sequenza completa di queste regioni del genoma precedentemente mancanti ci ha detto molto su come sono organizzate, cosa totalmente sconosciuta per molti cromosomi", ha detto Nicolas Altemose, un borsista post-dottorato presso l'Università della California, Berkeley.
Will Dunham
Reuters
Versione italiana Quotidiano Sanità
Ieri un consorzio di scienziati ha colmato la lacuna e in una ricerca pubblicata sulla rivista Science ha svelato anche quell’8% mancante. Il lavoro era già stato reso pubblico lo scorso anno ma mancava la sua revisione tra pari e la pubblicazione ufficiale.

Riepilogo dell'assemblaggio completo del genoma umano T2T-CHM13.
( A ) Ideogramma delle caratteristiche dell'assieme T2T-CHM13v1.1. Per ogni cromosoma (chr), vengono fornite le seguenti informazioni dal basso verso l'alto: lacune e problemi in GRCh38 risolti da CHM13 sovrapposti con la densità dei geni esclusivi di CHM13 in rosso; duplicazioni segmentali (SD) ( 42 ) e satelliti centromerici (CenSat) ( 30 ); e previsioni sull'ascendenza CHM13 (EUR, europea; SAS, dell'Asia meridionale; EAS, dell'Asia orientale; AMR, americana mista). La scala inferiore è misurata in Mbp. ( B e C ) Basi aggiuntive (non sinteniche) nell'assieme CHM13 relative a GRCh38 per cromosoma, con gli acrocentrici evidenziati in nero (B) e per tipo di sequenza (C). (Si noti che le annotazioni CenSat e SD si sovrappongono.) RepMask, RepeatMasker. ( D) Basi non gap totali nei rilasci del genoma di riferimento UCSC risalenti al settembre 2000 (hg4) e terminano con T2T-CHM13 nel 2021. Mt/Y/N, mitocondri, chrY e lacune. (Fonte: Science)
"La generazione di una sequenza del genoma umano veramente completa rappresenta un incredibile risultato scientifico, fornendo la prima visione completa del nostro progetto di DNA", ha affermato Eric Green, direttore del National Human Genome Research Institute (NHGRI), parte del National Institutes of Health degli Stati Uniti in una dichiarazione.
"Queste informazioni fondamentali rafforzeranno i numerosi sforzi in corso per comprendere tutte le sfumature funzionali del genoma umano, che a loro volta rafforzeranno gli studi genetici sulle malattie umane", ha aggiunto Green.
La versione completa è composta da 3.055 miliardi di coppie di basi, le unità da cui sono costruiti i cromosomi e i nostri geni, e 19.969 geni che codificano per le proteine.
Di questi geni, i ricercatori ne hanno identificati circa 2.000 nuovi. La maggior parte di questi è disabilitata, ma 115 potrebbero essere ancora attivi. Gli scienziati hanno anche individuato circa 2 milioni di varianti genetiche aggiuntive, 622 delle quali erano presenti in geni clinicamente rilevanti.
Il consorzio di scienziati che ha compiuto la ricerca è stato soprannominato Telomere-to-Telomere (T2T), dal nome delle strutture che si trovano alle estremità di tutti i cromosomi, la struttura filiforme nel nucleo della maggior parte delle cellule viventi che trasporta informazioni genetiche sotto forma di geni.
"In futuro, quando qualcuno avrà il proprio genoma sequenziato, saremo in grado di identificare tutte le varianti nel suo DNA e utilizzare tali informazioni per guidare meglio la propria assistenza sanitaria", ha detto Adam Phillippy, uno dei leader di T2T e ricercatore senior presso NHGRI.
"Completare davvero la sequenza del genoma umano è stato come indossare un nuovo paio di occhiali. Ora che possiamo vedere tutto chiaramente, siamo un passo più vicini alla comprensione di cosa significa", ha aggiunto Phillippy.
Tra le altre cose, le nuove sequenze di DNA hanno fornito nuovi dettagli sulla regione intorno a quello che viene chiamato centromero, dove i cromosomi vengono afferrati e separati quando le cellule si dividono per garantire che ogni cellula "figlia" erediti il numero corretto di cromosomi.
"Scoprire la sequenza completa di queste regioni del genoma precedentemente mancanti ci ha detto molto su come sono organizzate, cosa totalmente sconosciuta per molti cromosomi", ha detto Nicolas Altemose, un borsista post-dottorato presso l'Università della California, Berkeley.
Will Dunham
Reuters
Versione italiana Quotidiano Sanità
1 aprile 2022
© QS Edizioni - Riproduzione riservata
- Allegati
- La mappa